この記事で扱っていること
- 粒子解析の結果をラベル付きで表示する方法
を紹介しています。
注意:すべてのエラーを確認しているわけではないので、記事の内容を実装する際には自己責任でお願いします。また、エラー配線は適当な部分があるので適宜修正してください。
画像解析の目的の一つに、特定の対象物の数や(その画像上での)大きさや位置などの情報を調べる、粒子解析があります。
LabVIEWではVision Development Moduleのアドオンソフトを使うことでそのような解析も簡単に行えます。
やり方としては
- 画像を二値化する
- (必要に応じて)加工する
- 二値化後に「粒子」とみなせる部分に、解析したい内容を指定して粒子解析を実行する
といった関数を使うことになります。
この記事で紹介しているサンプルは、複数ある「粒子」に対する解析結果を表示するだけではなく、それぞれの解析結果がどの粒子についてなのかをという情報も画像上に粒子を番号付けして表示させています。
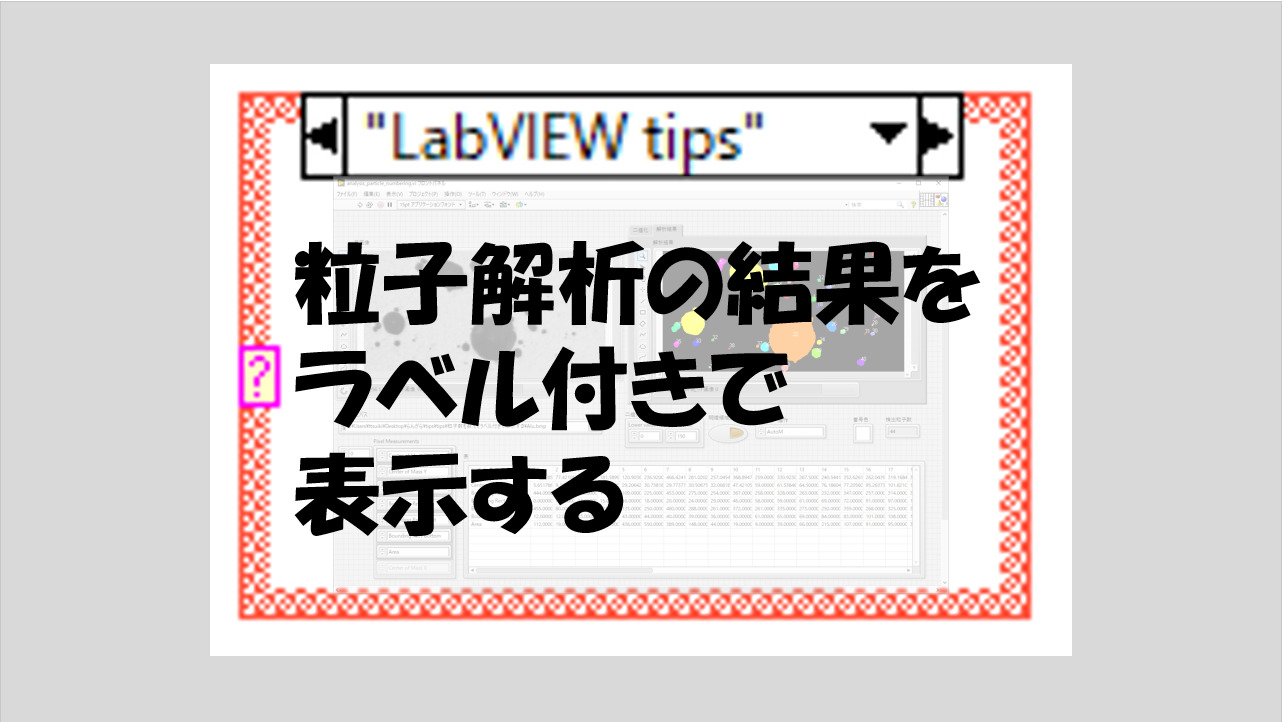
どんな結果になるか
フロントパネルには元々の画像を表示する以外に処理後の画像として二値化の結果および粒子解析の結果の画像を表示するようにしています。
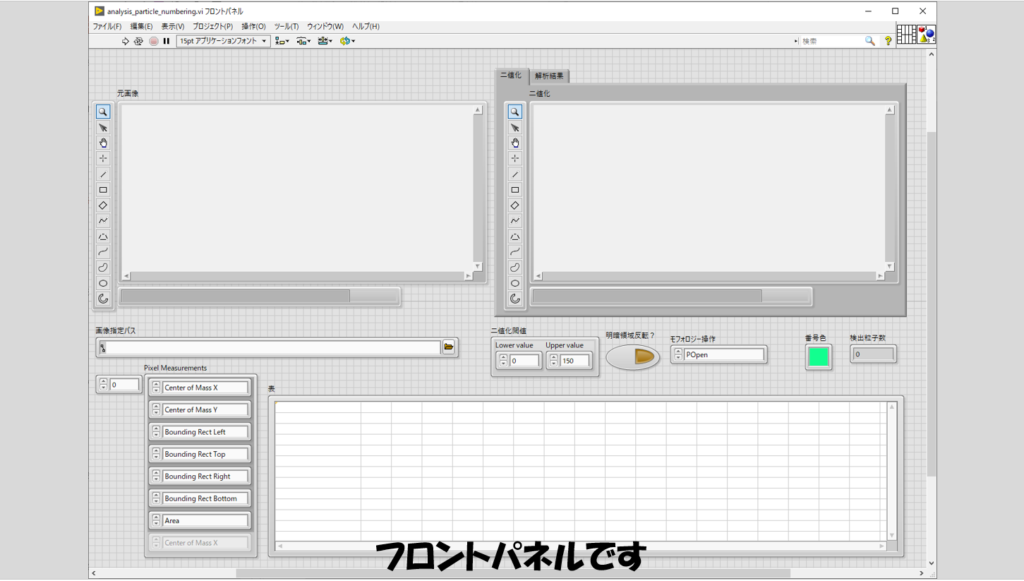
どの画像ファイルか、二値化の際のしきい値はどうするか、表示する番号の色はどうするか、そして解析はどのようなことを行うかを指定しています。
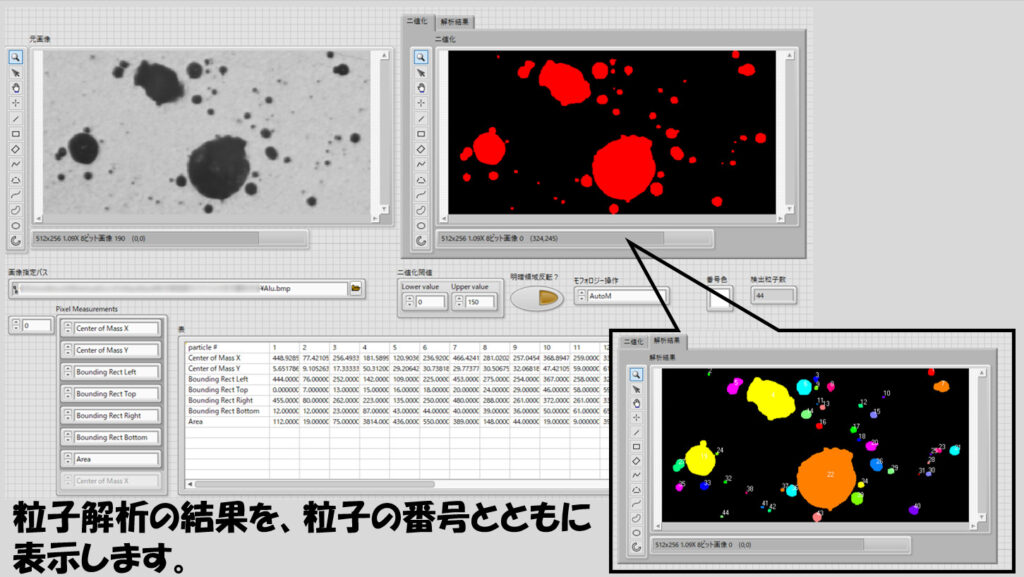
ただし、このプログラムでは、Center of Mass XとCenter of Mass Yが重要な解析指定パラメタになっていて、これらの情報によって各粒子に番号をつけています。(これらの情報が必要ない場合のプログラムは後半で扱います)
プログラムの構造
ブロックダイアグラムは以下のようにしています。
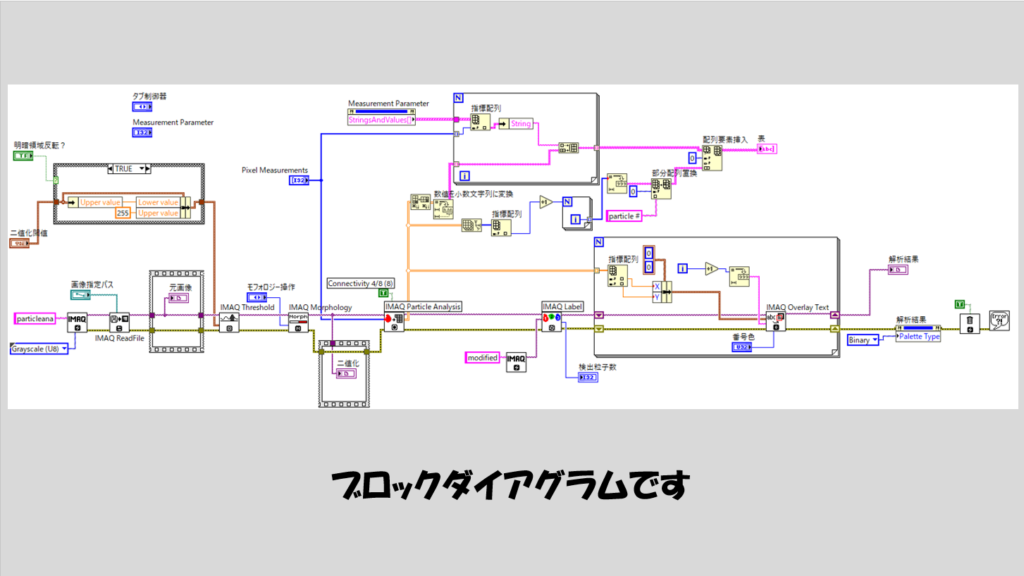
最初は、IMAQ Createで画像メモリ領域を用意し、IMAQ Read Fileで画像ファイルを読み込みます。この時点での画像情報、つまり解析前の元画像を確実に元画像表示器に反映させるためフラットシーケンスストラクチャを使用しています。
その後、IMAQ Thresholdで二値化を行っていますが、この時指定するパラメタである二値化閾値は必要に応じて反転させられるようにしています。
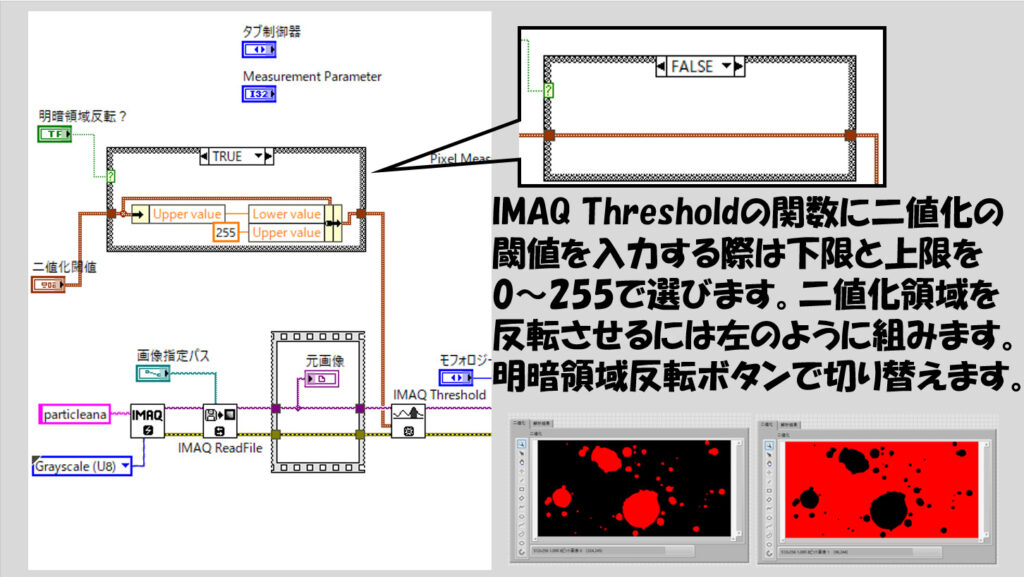
その後、これも必要に応じてですが、モフォロジー操作を行えるようにしています。必須の操作ではないので省いてもかまいません。(むしろ正しく操作しないと正しい結果が得られないこともあるので注意します)

その後はIMAQ Particle Analysisを行うことで二値化後に粒子とみなされた各対象について指定した解析方法を適用します。この結果をIMAQ Labelによって二値化領域の色分けをするという処理を挟んでForループに渡し、数字をテキストとしてオーバーレイしています。
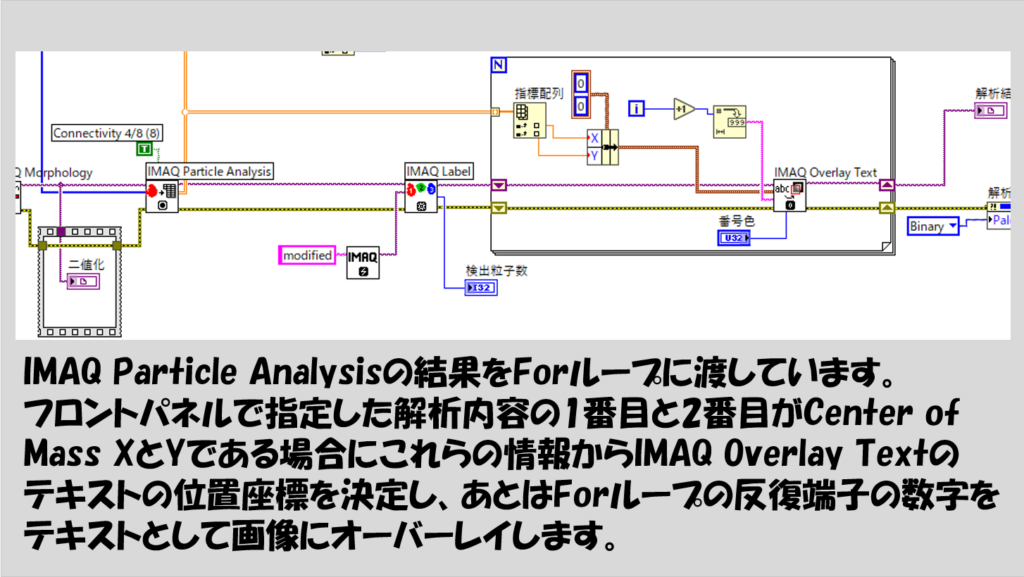
また、各解析結果をフロントパネルで見やすく表にするためにIMAQ Particle Analysisの結果をもとに表形式に情報をまとめているのが上部になります。
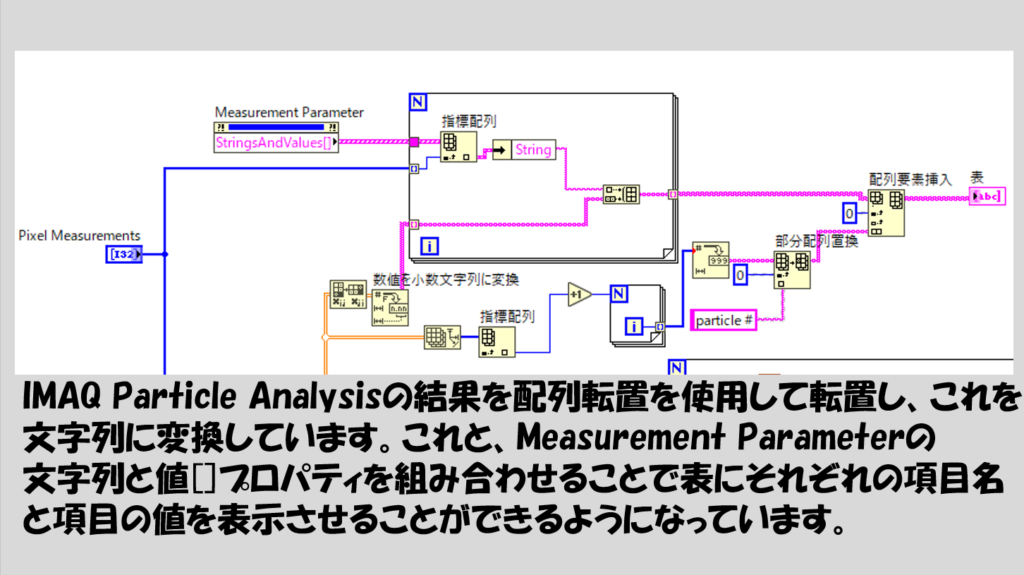
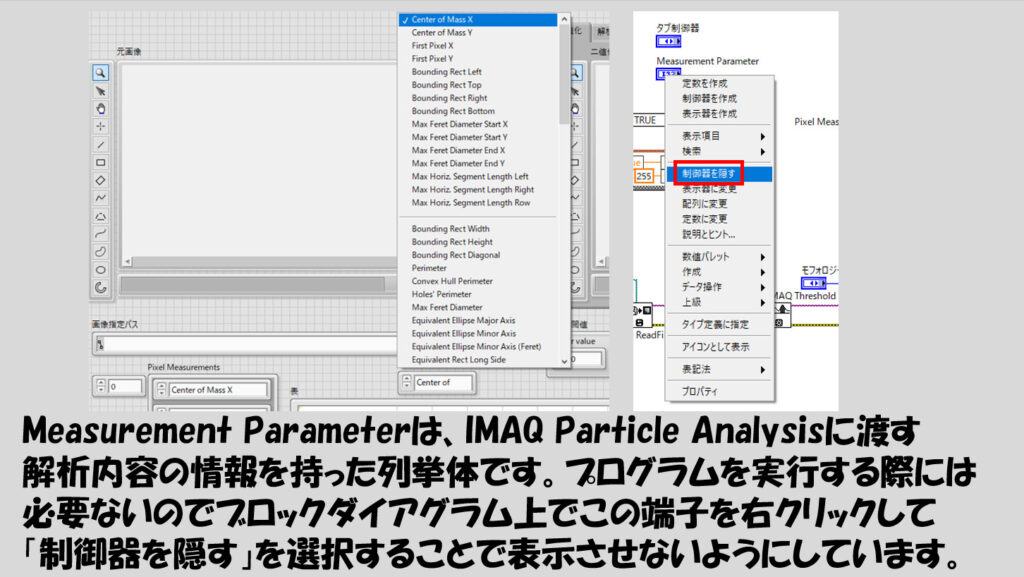
ちなみに、Measurement Parameterという端子に対してプロパティノードを使用していますが、これはフロントパネル上には表れていません。実行時には特に表示させている意味がないので隠しているためです。
このMeasurement Parameterは、Pixel Measurementsの配列の要素である列挙体となっています。(IMAQ Particle AnalysisのPixel Measurementsを右クリックして作成から定数を選ぶことでPixel Measurementsの配列は得られます)
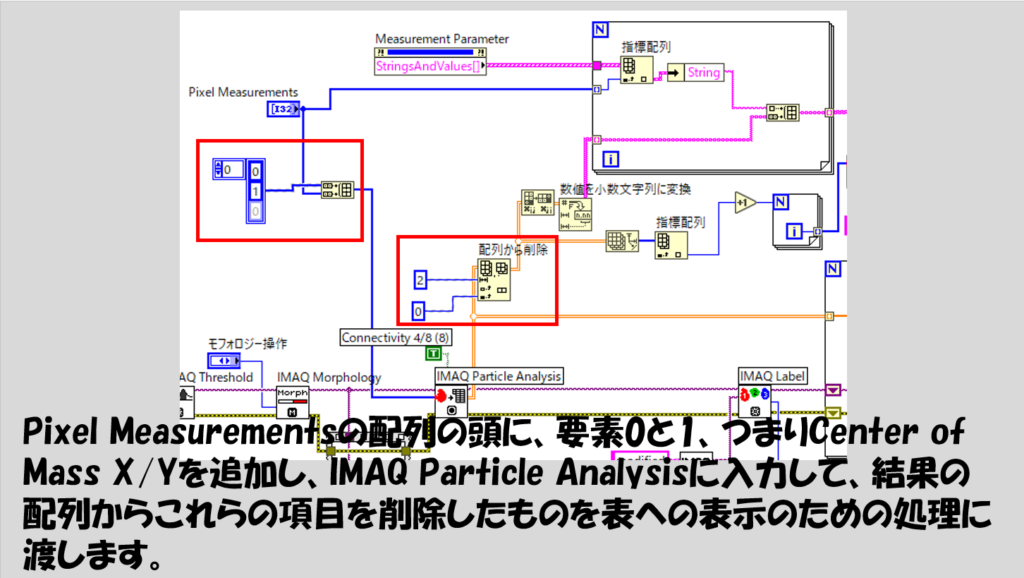
Center of Mass X/Yが必要ない場合
今回のプログラムで解析結果の画像上の各粒子に番号をつけているのはCenter of Mass X/Yの情報があるからというのは冒頭に紹介しました。
実際これらを指定しない場合、下記のように結果は正しく反映されません。
しかし、これらCenter of Mass X/Yが必要ないにもかかわらずわざわざ指定して表に表示させたくはない、という場合には、ブロックダイアグラムでは指定させるようにして、フロントパネルでは表示しないように組むことができます。
例えば以下のような方法が考えられます。
粒子解析については、Vision Development Moduleをインストールすることで付属するサンプルがあり、それらではまた今回の記事とは異なった方法でそれぞれの粒子の結果を表示することができます。ただ、同じ画像内の粒子の結果をまとめて表示するようなサンプルはなかったようなので今回のサンプルを考えてみました。参考にしていただければ嬉しいです。
ここまで読んでいただきありがとうございました。
コメント